Sobre • Metodologia • Inicialização • Conclusão • Créditos
A regulação da expressão gênica é um campo crucial da pesquisa científica, envolvendo diversos mecanismos que incluem o uso de pequenas moléculas de RNA, como siRNA e miRNA, para desempenhar um papel fundamental no silenciamento pós-transcricional de genes, conhecido como interferência por RNA (RNAi). A capacidade de utilizar esses tipos de RNA como ferramentas na criação de novas tecnologias tem despertado um interesse significativo entre os cientistas. Nesse contexto, apresentamos este algoritmo, projetado para automatizar o processo de encontrar siRNA, facilitando a pesquisa e o desenvolvimento de novas aplicações no campo da genética e biotecnologia.
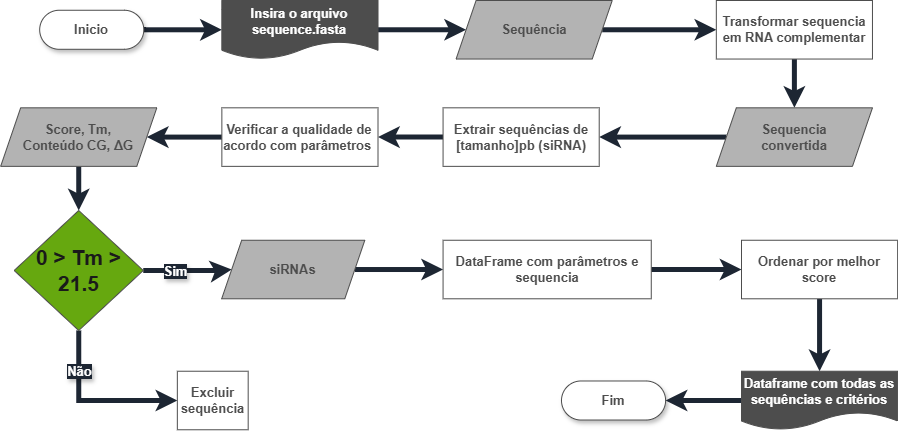
Para automatizar e aprimorar o processo de seleção de siRNA, desenvolvemos um algoritmo com as seguintes etapas:
📂Etapa 1: Entrada de Dados
O algoritmo recebe um arquivo FASTA inserido pelo usuário, contendo sequências de DNA a serem analisadas, onde cada sequência é identificada por um cabeçalho precedido por ">".
📂Etapa 2: Transcrição das Sequências de DNA
Para cada sequência de DNA no arquivo FASTA, o algoritmo realiza a transcrição, convertendo-a em uma sequência de RNA complementar.
📂Etapa 3: Identificação de siRNA
O algoritmo procura nas sequências de RNA resultantes por potenciais sequências de 22 pb que podem atuar como siRNA.
📂Etapa 4: Verificação da Qualidade do siRNA
De acordo com Reynolds et al. (2004), Ui-Tei et al. (2004) e Amarzguioui et al. estabeleceram critérios para determinar a funcionalidade das sequências de siRNA, que incluem o conteúdo de CG%, temperatura de melting e baixa estabilidade interna. Assim, os seguintes criterios seram usados para determinar se uma sequencia sera funcional. Os critérios são classificados em ordem de importância da seguinte forma:
| Rank | Critério | Pontuação |
|---|---|---|
| 1 | Ausência de repetições invertidas (hairpin) | - |
| 2 | Estabilidade interna baixa | -2 a 10 |
| 3 | Baixo teor de GC | 0 ou 4 |
| 4 | Posições específicas | -4 a 6 |
Assim, os siRNA candidatos são avaliados de acordo com esses critérios para determinar sua viabilidade. Para o primeiro critério, os siRNA que não atendem são descartados. Para os demais critérios, é atribuída uma pontuação: 10 pontos para o critério de estabilidade baixa, 4 ponto para o critério de baixo teor GC e 1 ponto para cada posição específicas.
É importante destacar que, no caso das posições específicas, cada posição que corresponde a um nucleotídeo ideal recebe 1 ponto, enquanto a não conformidade com essa posição retorna 0 pontos. Nas posições onde o nucleotídeo precisa ser específico, é deduzido 1 ponto se não estiver em conformidade.
A pontuação então sera trasformada em pencentual, onde apenas as sequencias com mais de 80% de conformidade seram levadas para as proximas fases.
📂Etapa 5: Exclusão de siRNA que não atendem aos critérios
Os siRNA que não atendem aos critérios de qualidade são excluídos da análise subsequente.
📂Etapa 9: Fim
O algoritmo conclui a análise das sequências de DNA, gerando uma tabela de resultados que pode ser utilizada para inferir a eficacia dos siRNA identificados, contribuindo para a pesquisa em biologia molecular e terapia genética.
O programa desenvolvido para a implementação do algoritmo de design de siRNA foi escrito em Python e utiliza diversas bibliotecas para processar e analisar as sequências de DNA. O Google Colab foi escolhido como plataforma para execução, permitindo fácil compartilhamento e colaboração. As principais bibliotecas utilizadas incluem Biopython para manipulação de sequências biológicas e Pandas para análise de dados. O código completo está disponível no repositório GitHub, permitindo que outros pesquisadores possam reproduzir e aprimorar o algoritmo conforme necessário.
Para inicializar o programa, siga os passos abaixo:
- Clone o repositório do GitHub:
git clone https://github.com/CatarinaRRF/IC_design_de_siRNA.git - Navegue até o diretório do projeto:
cd IC_design_de_siRNA - Execute o notebook no Google Colab ou localmente:
jupyter notebook siRNA_Design.ipynb
Esses passos permitirão que você configure o ambiente necessário para rodar o algoritmo e inicie o processo de design de siRNA.
Em conclusão, o algoritmo desenvolvido para o design de siRNA mostrou-se uma ferramenta eficaz e automatizada para a identificação de sequências de siRNA de alta qualidade. A aplicação dos critérios estabelecidos permitiu a seleção de candidatos viáveis, que foram posteriormente validados por meio de busca BLAST. Esses resultados destacam a importância do desenvolvimento de tecnologias automatizadas no campo da biotecnologia, facilitando a pesquisa e o desenvolvimento de novas terapias gênicas. Futuramente, planejamos desenvolver um aplicativo web baseado nesse algoritmo, permitindo que usuários de diversas áreas da ciência possam acessar e utilizar essa ferramenta de forma prática e intuitiva. Este aplicativo terá uma interface amigável e integrará todas as etapas do processo, desde a entrada de dados até a geração de resultados, contribuindo ainda mais para o avanço da pesquisa em interferência por RNA.
🌌 Acompanhe o desenvolvimento do aplicativo aqui
- Laboratório de Bioinformática e Análises Moleculares. Instituto de Genética e Bioquímica, Universidade Federal de Uberlândia, Campus Patos de Minas, Patos de Minas-MG
- Equipe de desenvolvimento: Catarina RRF, Valdeir de Paula e Matheus Souza